السلام عليكم ورحمة الله وبركاته
ورقة بحثية ممتازة اطلعت عليها مؤخرا، بعنوان:
ARE-mediated decay controls gene expression and cellular metabolism upon oxygen variations
منشورة فيه Nature Scientific Reports بتاريخ 26/03/2018 من جامعة بروكسل الحرة.
أكثر ما أعجبني في هذا البحث هو قواعد البيانات المستخدمة، وهي مهمة، لذلك أرغب بتوثيقها هنا.
____________________________________________________________________
اهتمامي بهذا البحث نابع لكوني أدرس بروتينات لها القدرة على الارتباط بمنطقة موجودة في عدد كبير من الـ mRNAs تسمى (AU-rich element (ARE، وارتباط البروتينات بهذه المنطقة يحفز تكسر الـ mRNAs بعملية تسمى (ARE-mediated decay (AMD.
معلومات إضافية عدم قراءتها لن يؤثر على فهمك للمقالة:
بعض الـ mRNAs تحتوى على ما يسمى بـ (AU-rich element (ARE في الـ 3′- Untranslated region. هي عبارة عن منطقة تحتوي على 50-150 نيوكليوتيد، وكما يوحي الاسم، غالب النيوكليوتيد فيها من نوع A & U.
غالبا يحتوي الـ mRNA على هذا الترتيب الخماسي AUUUA المكرر، وتم تصنيف الـ AREs إلى نوعين، بناء على عدد تكرار هذه المجموعة (AUUUA) وتوزيعها في الـ mRNAs، وصنفوا نوع ثالث لا يحتوي على هذا التكرار مطلقا، إنما فقط مناطق غنية بالقواعد النيتروجينية A&U.
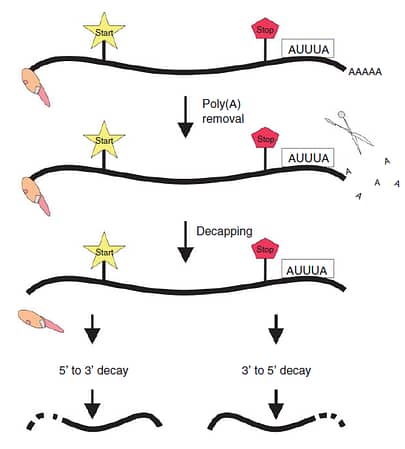
هذه المناطق التي نرمز لها بـ AREs تُعد مكان لإرتباط بعض البروتينات فيها (AUBP) وغالبا هذا الارتبطا يؤدي لتكسر الـ mRNA عبر عملية نسميها AMD (من المهم التنويه على أن هذه العملية تعتبر من العمليات الرئيسية المهمة لتنظيم التعبير الجيني). هذه العملية هي تكسر الـmRNA عبر deadenylation-dependent pathway وهي عبارة عن خطوتين:
- Exonucleolytic removal of the poly(A) tail.
- mRNA body degradation either by decapping followed by 5′ → 3′ degradation, or by continued exonucleolytic degradation in the 3′ → 5′ direction.
في هذا البحث تم التركيز على بروتين dTIS11 الذي ينتمي لعائلة TIS11/TTP في ذبابة الفاكهة، هذا البروتين له القدرة على الارتباط بـ ARE-containing mRNAs وتحفيز عملية AMD. رغِِب الباحثين بدراسة تأثير نقص الأكسجين الحاد “Hypoxia” على هذا البروتين، وعلى عملية الـ AMD، وبالتالي على الجينات التي يعمل بروتين dTIS11 عليها.
نتائج الدراسة التركيز على القواعد البيانية المستخدمة + التجارب والأجهزة المميزة:
1- تم تعريض خلايا ذبابة الفاكهة S2 إلى نقص الأكسجين الحاد (تركيز 1%) لمدة 18 ساعة، ثم رصد التعبير الجيني لبروتين dTIS11 . بشكل عام، نقص الأكسجين يسبب نقص كبير في الطاقة ATP وبالتالي توقف العمليات التي تستهلك كمية طاقة كبيرة، ومنها عملية تصنيع البروتين mRNA translation، وهذا أيضا ينطبق على بروتين dTIS11، مستوى الـ mRNA لم يتغير، بينما مستوى البروتين قل بشكل كبير جدا مما يعكس التكسر السريع للبروتين، وتوقف عملية الـ translation.
These results reveal that dtis11 expression is strongly and dynamically regulated by oxygen concentration, thereby suggesting that dTIS11-dependent AMD may contribute to gene expression reprogramming upon variations in oxygen levels.
2- عمل مسح كامل للـ mRNA في حالة نقص الأكسجين في الخلية (1%)، وفي الحالة الطبيعية (21%)، مع تصنيفها بحسب الـ ARE-score لمعرفة الـ mRNAs المعرضة لـ AMD:
* هذه هي الجزئية المهمة جدا في الورقة بالنسبة لي.
-> تم تعريض مجموعة من خلايا S2 لمستوى أكسجين ناقص (1%) و مجموعة لمستوى طبيعي (21%).
-> بعد 18 ساعة تم استخلاص كل الـ mRNA في الخلايا، وعمل RNA-seq، ووجدوا أن 695 جين كانوا مرتفعين في حالة نقص الاكسجين مقارنة بالخلايا الطبيعية، و 456 جين انخفض تعبيرهم الجيني.
-> الآن يرغبون بتصنيف هذه الـ (mRANs (transcripts بناء على وجود الـ ARE motifs فيها، تم حساب الـ AREScores لكل الجينات هذه، ولكل الـ isoforms المسجلة في NCBI RefSeq mRNA database !!
السؤال: ماهو الـ AREScore؟ كيف يعمل، وكيف يُحسب؟
الجواب: هي عبارة عن أداة تم تصميمها من قِبل مجموعة باحثين في مركز أبحاث السرطان الألماني بالتعاون مع باحث من معهد المعلوماتية في جامعة LMU في ألمانيا. أُطلقت رسميا في عام 2012.
فكرتها أنه بعد إدخال mRNAs sequences يخرج لك البيانات على هيئة نقاط رقمية (مثلا 1 أو 2..إلخ)، لكل Transcripts، ولكن، كيف يحسبون هذه النقاط؟
هذه الصورة توضح الحسبة:

الـ Total score هي مجموع:
1- عدد الـ AUUUA pentamer في الـ mRNA.
2- موقع الـ AUUUA pentamer (كلما كانوا متقاربين، حازوا نقاط أكثر).
3- موقع الـ AU-blocks ( هل الـ AUUUA pentamer موجود في مواقع غنية بالـ AU nucleotides؟) إذا نعم، أضِف نقطة!
4- طول الـ 3′-UTR. الـ AREScore تُحسب في حال كان طول هذه المنطقة اكثر من أو تساوي 10nt.
طبعا بإمكان المستخدم يعدل ويغير من هذه المعايير حسب احتياجه ورغبته.
هذا هو رابط موقع الأداة: http://arescore.dkfz.de/arescore.pl
______________
نعود الآن للبحث، استخدموا هذه الاداة لتصنيف كل الجينات بحسب الـ AREScore، طبعا اعتمدوا على أنه عندما تكون قيمة الـ AREScore ≥ 2 وقتها تُعد منطقة الـ ARE قابلة لأن ترتبط البروتينات بها وتحفز الـ AMD، أقل من 2 تعتبر mRNAs غبر معرضة لعملية AMD.
الآن، أصبح لدينا مجموعتين من الجينات، أحدها مع AREScore≥ 2 والأخرى AREScore < 2، صنفها الباحثين أكثر بناء على ارتفاع/ انخفاض التعبير الجيني أثناء نقص الأكسجين، والنتيجة:
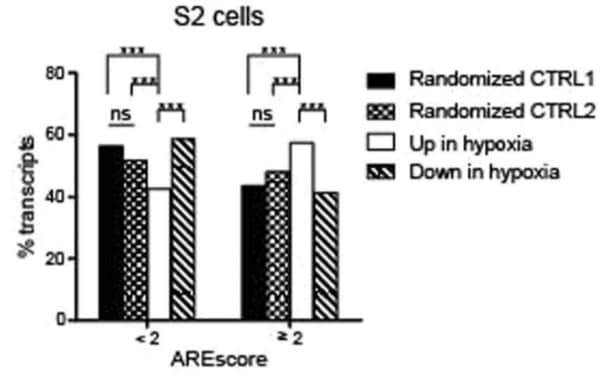
الملاحظة المُستنتجة:
- أكثر الجينات التي ارتفع التعبير الجيني لها في حالة نفص الأكسجين تتبع لمجموعة AREScore≥ 2.
بقدرتنا الآن تحليل هذه المعلومات، علما أن التحليل لا يعني الصحة، إنما هو نتيجة ربط البيانات السابقة ببعضها، واستنتاج مبدئي يحتاج لتجارب عملية لتثبيته واقعيا:
قلنا أن منطقة الـ ARE يرتبط بها بروتينات مثل dTIS11 ويعملون على تحفيز تكسرها عبر عملية تسمى AMD، وعندما يكون الـ AREScore أكبر أو يساوي 2، يعني أن للبروتينات القدرة على الإرتباط، وبالتالي تفعيل AMD.
وقلنا سابقا أيضا أن dTIS11 انخفض بشكل كبير جدا أثناء نقص الأكسجين.
وقلنا أخيرا أن كثير من الجينات التي تحتوي على ARE ارتفع التعبير الجيني لها أثناء نقص الأكسجين.
كل هذا يدفعنا لاستنتاج أن البروتين dTIS11 كان يعمل على هذه البروتينات ويحفز تكسرها في حالة الأكسجين الطبيعي، ولكن عند نقص الأكسجين، ونقص عدد هذا البروتين، استقر التعبير الجيني لهذه الجينات وارتفع.
ولاختبار هذا الإستنتاج، ننتقل إلى التجربة الثالثة التي تمت في هذا البحث.
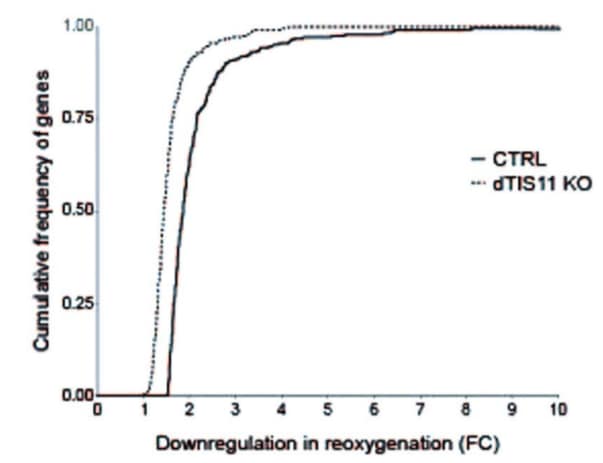
3- في هذه التجربة رغب الباحثين في معرفة ما إن كان الإنخفاض في التعبير الجيني لـ ARE-containing mRNA بعد الـ reoxygenation كان بسبب بروتين الـ dTIS11 أم لا (بسبب تحفيزه لعملية AMD).
لمعرفة ذلك، صمم الباحثين Crispr-Cas يستهدف جين dTIS11 لتثبيطه، والبيانات النهائية لهذه التجربة تفيد أن بعض الجينات ارتفع تعبيرها الجين حينما تم تثبيط dTIS11. أيضا، وجدوا أن هذه الجينات تلعب دورا في العمليات الأيضية، وبالتالي dTIS11 بدوره يعتبر بروتين منظم مهم لعمليات التكيف الأيضية استجابة للتغير في مستوى الأكسجين في الخلية.
يهمني أكلمكم عن البرنامج/الموقع اللي استخدموه لتحليل بيانات الـ RNA-seq والتغير في التعبير الجيني، وأيضا باستخدامه صنفوا الجينات حسب الوظائف، ورسم لهم MA plot و MDS plot وأشياء كثيرة بعضها ما اكتشفتها حتى الآن، لكن واضح أنه متعوب عليه، هذا الموقع يسمى Degust، لكن ما تقدرون تستخدمونه فعليا إلا لو عندكم بيانات من جهاز RNA-seq لأن عمله ببساطة هو تحليلها، للفضوليين مثلي، الموقع أعطانا تجربة، صفحة فيها بيانات وممكن تحوس فيها ليين تتعلم وتعرف، بصراحة عمل جباااار، هنا الموقع الرسمي:
وهنا التجربة:
http://degust.erc.monash.edu/degust-old/compare.html?code=example
_______________________
الورقة ما انتهت، فيها تفاصيل أكثر، والبحث مب سهل أبدا، الباحثين عملوا على فكرتهم من جهات كثيرة، لكن نقلت هنا المهم واللي أنا شخصيا بأحتاج أرجع له أكثر من مرة.
هنا ملخص مناقشة الورقة “بالإنجليزية”:
- dTIS11 protein level significantly decreased upon hypoxia, due to overall mRNA translation blockade (as ATP level decreased) as well as protein degradation by ubiquitin-independent degradation by the 20 S proteasome.
- The response of hypoxia-tolerant systems to oxygen deprivation occurs in two phases that can be considered as defense and rescue processes. The first lines of defense against hypoxia include a balanced suppression of ATP demand and ATP supply pathways; this regulation stabilizes adenylate concentrations at new steady-state levels as ATP turnover rates greatly decline. Energy-consuming processes such as ion pumping and protein synthesis are down-regulated. The latter process results from a rapid and massive polysome disassembly leading to a translational arrest. Te secondary rescue mechanisms include adaptation of the metabolic program and rely on major changes in the gene expression program mainly under control of the HIF family of transcription factors.
- “Observations support the idea that dTIS11 controls the AMD of many proteins. However, during hypoxia, and because of dTIS11 absence, many ARE-containing mRNAs are survived and successfully translated, many of these proteins are involved in hypoxia-related metabolic processes and they are important for cellular adaption.”
- Researchers went far by further studying ImpL3 gene, which encodes lactate dehydrogenase (LDH) in Drosophila, AREScore= 4.1. It’s been shown that this protein is immediately degraded upon reoxygenation in a dTIS11-dependent manner. In dTIS11 deficient cells upon reoxygenation, ImpL3 and LDH accumulate, as so, this will impair the recovery of oxidative phosphorylation and cell proliferation processes.
-
In conclusion, our study identifes ARE-mediated decay as a new regulatory mechanism contributing to gene expression reprogramming during normoxia/hypoxia transitions and suggests that global inhibition of protein synthesis infuences the expression of specifc gene subsets containing ARE by modulating the level of the post-transcriptional regulator dTIS11.”
_______________________
تحديث:
وجدت هذا الموقع (AREsite2):
http://nibiru.tbi.univie.ac.at/AREsite2/welcome
فيما يخص AREScore، وهو موقع مُحدث أكثر، ويشمل مواقع أكثر من 3′-UTR، وأيضا AU, GU and U-rich elements. وفي النتائج يعطي الأبحاث التي درست هذه المنطقة في الجين (رابط يحولك لـ Pubmed). تم تطوير الموقع من باحثين مختلفين، من جامعة فينا في النمسا. وطبعا، أفضل من السابق ذكره بكثير وبدون مقارنة.
للمهتمين، هذه المراجع:
Research article: https://doi.org/10.1038/s41598-018-23551-8
The role of AUF1 in regulated mRNA decay: https://doi.org/10.1002/wrna.26
Genome-Wide Assessment of AU-Rich Elements by the AREScore Algorithm:
AREsite2: an enhanced database for the comprehensive investigation of AU/GU/U-rich elements: https://doi.org/10.1093/nar/gkv1238

218 رد على “تحديث: بحث علمي: mRNA destabilization during hypoxia via ARE-mediated decay”
Hello there, You have done a great job. I’ll definitely digg it and personally suggest to my friends.
I am sure they’ll be benefited from this web site.
I am really enjoying the theme/design of your web site.
Do you ever run into any browser compatibility issues?
A handful of my blog readers have complained about my blog not operating correctly in Explorer but looks great in Firefox.
Do you have any tips to help fix this problem?
Join our affiliate family and watch your profits soar—sign up today!
Start profiting from your traffic—sign up today!
Earn up to 40% commission per sale—join our affiliate program now!
Monetize your traffic with our affiliate program—sign up now!
Turn your traffic into cash—join our affiliate program!
Fast indexing of website pages and backlinks on Google https://is.gd/r7kPlC
Earn your airdrop on Aster https://is.gd/ZceEI6
https://shorturl.fm/vXjHo
https://shorturl.fm/5Rg04
https://shorturl.fm/zLfJs
https://shorturl.fm/LCpEc
https://shorturl.fm/72j0E
https://shorturl.fm/D7Zlk
https://shorturl.fm/Cg6DZ
https://shorturl.fm/DT19e
https://shorturl.fm/GD21Q
https://shorturl.fm/rOWdi
https://shorturl.fm/EpY4r
https://shorturl.fm/IZnQu
https://shorturl.fm/ZXlqU
https://shorturl.fm/G00RP
marque kamagra made in usa
commander kamagra générique
koupit kamagra v kanadě
pouze doručení kamagra
buy itraconazole uk in store
buy itraconazole price australia
get fildena canada purchase
get fildena purchase usa
buying gabapentin low price
buy gabapentin cheap genuine
purchase flexeril cyclobenzaprine uk meds
buy cheap flexeril cyclobenzaprine buy in london
buy dutasteride generic equivalent
how to get a doctor to prescribe dutasteride
buy avodart uk over the counter
how to order avodart generic compare
ordering staxyn cheap no prescription
purchase staxyn american express
xifaxan online consultation
discount xifaxan uk order
ordering rifaximin no prescription overnight delivery
buy rifaximin generic ireland
enclomiphene shipped overnight no perscription
get enclomiphene canadian discount pharmacy
how to order androxal price uk
order androxal canada drugs
https://shorturl.fm/NdQI3
https://shorturl.fm/9VjPN
https://shorturl.fm/JEYnp
https://shorturl.fm/XM8mL
https://shorturl.fm/Mlvuf
https://shorturl.fm/b5ozc
https://shorturl.fm/5obAT
https://shorturl.fm/xpBbN
https://shorturl.fm/SV8Vz
https://shorturl.fm/ycLcu
https://shorturl.fm/ASSfH
https://shorturl.fm/QeZrp
https://shorturl.fm/V7cLd
https://shorturl.fm/ZOujd
https://shorturl.fm/mrvpz
https://shorturl.fm/Ss26n
https://shorturl.fm/mO7L5
https://shorturl.fm/yayx8
https://shorturl.fm/0uZ81
https://shorturl.fm/kXPub
https://shorturl.fm/KwkMo
https://shorturl.fm/maDIZ
https://shorturl.fm/Kcgsz
https://shorturl.fm/UiwF5
https://shorturl.fm/vGEat
https://shorturl.fm/04D9i
https://shorturl.fm/J1koR
https://shorturl.fm/y8hX4
https://shorturl.fm/DcMGY
https://shorturl.fm/0wbS1
https://shorturl.fm/9nO0U
https://shorturl.fm/FHUXb
https://shorturl.fm/WXaDN
https://shorturl.fm/egZvR
https://shorturl.fm/egZvR
https://shorturl.fm/rRFVQ
https://shorturl.fm/YuI97
https://shorturl.fm/CYVDQ
https://shorturl.fm/85whJ
https://shorturl.fm/wZVHT
https://shorturl.fm/rECMy
https://shorturl.fm/p7OHS
https://shorturl.fm/jHJNV
https://shorturl.fm/pP2oj
https://shorturl.fm/30Uv1
https://shorturl.fm/2yttT
Become our affiliate—tap into unlimited earning potential! https://shorturl.fm/TE8TW
Refer friends, collect commissions—sign up now! https://shorturl.fm/1Aahh
Refer friends, collect commissions—sign up now! https://shorturl.fm/IDd4J
Earn passive income on autopilot—become our affiliate! https://shorturl.fm/K510y
Boost your income effortlessly—join our affiliate network now! https://shorturl.fm/qiY64
Monetize your traffic with our affiliate program—sign up now! https://shorturl.fm/9UAbs
Sign up and turn your connections into cash—join our affiliate program! https://shorturl.fm/w0Rb7
Boost your income—enroll in our affiliate program today! https://shorturl.fm/kJsUO
Sign up for our affiliate program and watch your earnings grow! https://shorturl.fm/XdjLO
Sign up and turn your connections into cash—join our affiliate program! https://shorturl.fm/IUgbT
Boost your profits with our affiliate program—apply today! https://shorturl.fm/dejgk
Tap into unlimited earnings—sign up for our affiliate program! https://shorturl.fm/xU1E1
Tap into unlimited earnings—sign up for our affiliate program! https://shorturl.fm/0MsOe
Become our affiliate—tap into unlimited earning potential! https://shorturl.fm/6q2Pj
Turn your audience into earnings—become an affiliate partner today! https://shorturl.fm/7p2oo
Maximize your income with our high-converting offers—join as an affiliate! https://shorturl.fm/GwXnd
Your audience, your profits—become an affiliate today! https://shorturl.fm/1psgs
Promote our products and earn real money—apply today! https://shorturl.fm/OA0vU
Drive sales and watch your affiliate earnings soar! https://shorturl.fm/ruwJK
Start earning every time someone clicks—join now! https://shorturl.fm/aGQOW
Join our affiliate program and watch your earnings skyrocket—sign up now! https://shorturl.fm/t1pBh
Promote, refer, earn—join our affiliate program now! https://shorturl.fm/aOgWF
Earn recurring commissions with each referral—enroll today! https://shorturl.fm/TjuHP
Start earning passive income—become our affiliate partner! https://shorturl.fm/VwDXO
Get started instantly—earn on every referral you make! https://shorturl.fm/qtNqc
Turn your audience into earnings—become an affiliate partner today! https://shorturl.fm/55M23
Share our link, earn real money—signup for our affiliate program! https://shorturl.fm/4WXSy
Get paid for every click—join our affiliate network now! https://shorturl.fm/4g4Pt
Monetize your audience—become an affiliate partner now! https://shorturl.fm/i8PuL
Monetize your audience—become an affiliate partner now! https://shorturl.fm/fLk9T
Unlock exclusive rewards with every referral—enroll now! https://shorturl.fm/v4que
Start earning on every sale—become our affiliate partner today! https://shorturl.fm/hD1a3
Start earning instantly—become our affiliate and earn on every sale! https://shorturl.fm/XMb8H
Sign up now and access top-converting affiliate offers! https://shorturl.fm/5Lmmd
Share our products, earn up to 40% per sale—apply today! https://shorturl.fm/y3Czo
Monetize your influence—become an affiliate today! https://shorturl.fm/mBWrp
Monetize your influence—become an affiliate today! https://shorturl.fm/EYr8e
Get paid for every referral—sign up for our affiliate program now! https://shorturl.fm/qTpFD
Дюна – Свет звезды скачать песню и слушать онлайн
https://allmp3.pro/2650-djuna-svet-zvezdy.html
Virus Sasha First, R Max – Ты Меня Не Ищи (Dj Baur 22 Reboot) скачать песню и слушать онлайн
https://allmp3.pro/3175-virus-sasha-first-r-max-ty-menja-ne-ischi-dj-baur-22-reboot.html
SAMURAYKI – ВХЛАМ скачать песню и слушать онлайн
https://allmp3.pro/3203-samurayki-vhlam.html
Макс Барских – Неслучайно скачать песню и слушать онлайн
https://allmp3.pro/2578-maks-barskih-nesluchajno.html
Юрий Антонов – У берез и сосен скачать песню и слушать онлайн
https://allmp3.pro/2558-jurij-antonov-u-berez-i-sosen.html
Александр Добрынин – Молодая мама скачать песню и слушать онлайн
https://allmp3.pro/2618-aleksandr-dobrynin-molodaja-mama.html
Юрий Антонов – От печали до радости скачать песню и слушать онлайн
https://allmp3.pro/2507-jurij-antonov-ot-pechali-do-radosti.html
Captain Max – Я Запомню скачать песню и слушать онлайн
https://allmp3.pro/2861-captain-max-ja-zapomnju.html
Pharaon – Халливуд Хоус скачать песню и слушать онлайн
https://allmp3.pro/2906-pharaon-hallivud-hous.html
Мария Маевская – Влюбиться Навсегда скачать песню и слушать онлайн
https://allmp3.pro/3010-marija-maevskaja-vljubitsja-navsegda.html
https://shorturl.fm/TDuGJ
https://shorturl.fm/Kp34g
https://shorturl.fm/JtG9d
https://shorturl.fm/fSv4z
https://honda-fit.ru/forums/index.php?autocom=gallery&req=si&img=7130
https://vitz.ru/forums/index.php?autocom=gallery&req=si&img=4909
http://wish-club.ru/forums/index.php?autocom=gallery&req=si&img=5433
http://terios2.ru/forums/index.php?autocom=gallery&req=si&img=4795
https://mazda-demio.ru/forums/index.php?autocom=gallery&req=si&img=6525
https://mazda-demio.ru/forums/index.php?autocom=gallery&req=si&img=6392
https://honda-fit.ru/forums/index.php?autocom=gallery&req=si&img=7287
https://hrv-club.ru/forums/index.php?autocom=gallery&req=si&img=7141
http://wish-club.ru/forums/index.php?autocom=gallery&req=si&img=5303
https://mazda-demio.ru/forums/index.php?autocom=gallery&req=si&img=6500
La Vtornik feat. Daniel Shake & Керил – Миллиарды скачать бесплатно mp3 и слушать онлайн https://shorturl.fm/qtipo
Александр Берег – Не Пойманные Ветром скачать песню и слушать онлайн https://shorturl.fm/W739w
Александр Сергеев – Дежавю скачать песню и слушать бесплатно https://shorturl.fm/kIgRc
Samurayki – Вхлам скачать и слушать онлайн https://shorturl.fm/9cHnD
Елена Ваенга Feat. & Алена Петровская – Обыкновенный скачать песню бесплатно в mp3 и слушать онлайн https://shorturl.fm/y8eAq
Xcho – Ты и Я скачать и слушать песню https://shorturl.fm/DpAPJ
Елена Ваенга Feat. & Алена Петровская – Обыкновенный скачать песню бесплатно в mp3 и слушать онлайн https://shorturl.fm/y8eAq
Игорь Balan – Веточка Сирени скачать и слушать песню https://shorturl.fm/CGXMg
https://shorturl.fm/Kp34g
https://shorturl.fm/IPXDm
https://shorturl.fm/DA3HU
https://shorturl.fm/nqe5E
https://shorturl.fm/xlGWd
https://shorturl.fm/hQjgP
https://shorturl.fm/a0B2m
https://shorturl.fm/oYjg5
Илона Истомина – Амор скачать и слушать mp3 https://shorturl.fm/J61Qo
Shoo – Около скачать песню в mp3 и слушать онлайн https://shorturl.fm/82vM7
Комбинация – Бухгалтер скачать песню и слушать онлайн https://shorturl.fm/ZX9rh
https://shorturl.fm/5JO3e
Semikrasov – Зашивать скачать и слушать онлайн https://shorturl.fm/FGqmJ
Никита Киоссе – Не В Себе скачать mp3 и слушать онлайн бесплатно https://shorturl.fm/UMne5
Nezvonimne – Мани Манят скачать песню в mp3 и слушать онлайн https://shorturl.fm/oSLvJ
Алмас Багратиони – Доброту За Деньги Не Купить скачать mp3 и слушать бесплатно https://shorturl.fm/1XJlc
Моя Мишель feat. Dose – Пташка скачать mp3 и слушать онлайн бесплатно https://shorturl.fm/uhOR3
Mosovich – Помогая скачать песню на телефон и слушать бесплатно https://shorturl.fm/b8m1w
Таисия Повалий – Губы твои алые скачать песню бесплатно в mp3 и слушать онлайн https://shorturl.fm/m88VY
https://shorturl.fm/9fnIC
https://shorturl.fm/5JO3e
https://shorturl.fm/A5ni8
https://shorturl.fm/FIJkD
https://shorturl.fm/A5ni8
https://shorturl.fm/5JO3e
https://shorturl.fm/TbTre
https://shorturl.fm/FIJkD
https://shorturl.fm/m8ueY
https://shorturl.fm/m8ueY
https://shorturl.fm/6539m
https://shorturl.fm/a0B2m
https://shorturl.fm/m8ueY
https://shorturl.fm/FIJkD
Awesome https://shorturl.fm/oYjg5
https://honda-fit.ru/forums/index.php?autocom=gallery&req=si&img=7238
http://terios2.ru/forums/index.php?autocom=gallery&req=si&img=4668
http://toyota-porte.ru/forums/index.php?autocom=gallery&req=si&img=3298
https://hrv-club.ru/forums/index.php?autocom=gallery&req=si&img=6976
https://myteana.ru/forums/index.php?autocom=gallery&req=si&img=6653
http://wish-club.ru/forums/index.php?autocom=gallery&req=si&img=5268
https://vitz.ru/forums/index.php?autocom=gallery&req=si&img=4849
https://mazda-demio.ru/forums/index.php?autocom=gallery&req=si&img=6367
https://hrv-club.ru/forums/index.php?autocom=gallery&req=si&img=6884
https://hrv-club.ru/forums/index.php?autocom=gallery&req=si&img=7179
Олег Газманов – Доля скачать песню и слушать онлайн https://shorturl.fm/20kTg
Бауырлар тобы – Гулмира скачать и слушать онлайн https://shorturl.fm/9hKZj
Звонкий – Голоса скачать mp3 и слушать онлайн https://shorturl.fm/8oQH3
Mary Gu – Астероид (Denis Bravo Remix) скачать песню бесплатно в mp3 и слушать онлайн https://shorturl.fm/rHPhk
Dinar Rahmatullin – Мама скачать бесплатно mp3 и слушать онлайн https://shorturl.fm/nho0u
MITCHEL – А уже фсё скачать песню бесплатно в mp3 и слушать онлайн https://shorturl.fm/zviO5
Артик и Асти – Все мимо, а-а скачать бесплатно и слушать онлайн https://shorturl.fm/aptw0
Admood – Счастлив скачать песню на телефон и слушать бесплатно https://shorturl.fm/SbB7H
https://vitz.ru/forums/index.php?autocom=gallery&req=si&img=4806
https://myteana.ru/forums/index.php?autocom=gallery&req=si&img=6921
https://vitz.ru/forums/index.php?autocom=gallery&req=si&img=5098
https://honda-fit.ru/forums/index.php?autocom=gallery&req=si&img=7140
https://myteana.ru/forums/index.php?autocom=gallery&req=si&img=6708
https://myteana.ru/forums/index.php?autocom=gallery&req=si&img=6695
https://myteana.ru/forums/index.php?autocom=gallery&req=si&img=6695
http://toyota-porte.ru/forums/index.php?autocom=gallery&req=si&img=3348
http://wish-club.ru/forums/index.php?autocom=gallery&req=si&img=5392
http://passo.su/forums/index.php?autocom=gallery&req=si&img=4293
http://wish-club.ru/forums/index.php?autocom=gallery&req=si&img=5488
https://mazda-demio.ru/forums/index.php?autocom=gallery&req=si&img=6599
https://myteana.ru/forums/index.php?autocom=gallery&req=si&img=6611
http://wish-club.ru/forums/index.php?autocom=gallery&req=si&img=5233